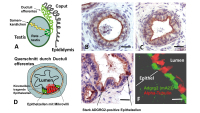
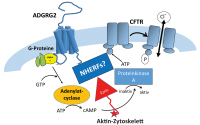
ADGRG2-Sequenz
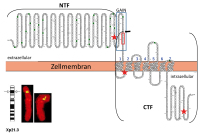
Abbildung 2: ADGRG2-Sequenz und X-chromosomale Lokalisation. Die etwa 1000 Aminosäuren lange Proteinkette
ist durch Kreise im Einbuchstaben-Code abgebildet, Splice-Varianten sind nicht dargestellt; mögliche
N-Glykosylierungsstellen sind grün unterlegt. Wie alle Mitglieder der aGPCR-Familie hat ADGRG2 einen
langen extrazellulären N-Terminus, eine zentrale 7TM-Region (Membran-spannende Helices, durch blaue
Ziffern markiert) sowie einen intrazellulären C-Terminus. Die Proteinkette ist in 2 Untereinheiten gespalten
(durch schwarze Klammern eingerahmt), als N-terminales Fragment (NTF) und C-terminales Fragment (CTF)
bezeichnet; der schwarze Pfeil markiert die Spaltstelle. Die GAIN-Domäne (blauer Kasten) katalysiert die
Autoproteolyse. Dabei wird die sogenannte Stachel-Sequenz (roter Kasten) freigelegt. Die Positionen der
3 proteinverkürzenden LoF-Mutationen [12] sind durch rote Sterne hervorgehoben. Sie rufen den gleichen
Phänotyp hervor, obwohl die Proteinkette an ganz unterschiedlichen Stellen abbricht. Anscheinend reicht
allein der Ausfall der letzten 30 Aminosäuren bereits zum vollständigen Verlust der Rezeptor-Funktion.
© C. Kirchhoff
Keywords:
ADGRG2,
Schema,
X-Chromosom